
Capítulo 7 Manipulación de datos ordenados usando dplyr y tidyr II
A esta altura del libro vimos varios conjuntos de datos. Cada uno tiene una pinta un poco distinta y trabajar con ellos presenta distintos desafíos. En este capítulo veremos como manipular los datos para organizarlos de distinta manera y que nos sirvan para resolver distintos problemas.
Pero primero algunas definiciones.
Cuando hablamos de datos “ordenados” , o “tidy”, o en formato “largo”, nos referimos a aquellos set de datos en los cuales:
- cada fila es una observación
- cada columna es una variable
Los datos en formato “ancho” pueden ser muy variados, por lo que son un poco más complejos de definir. Pero la idea general es que:
- cada fila es un “item”
- cada columna es una variable
- muchas veces los nombres de las columnas son variables de los datos
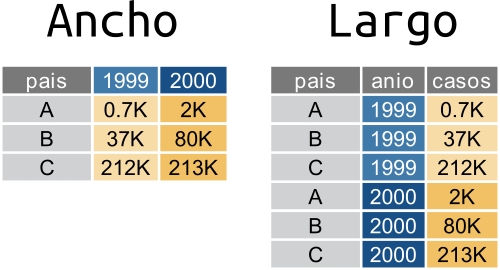
Una tabla en formato largo va a tener una cierta cantidad de columnas que cumplen el rol de identificadores y cuya combinación identifican una única observación y una única columna con el valor de la observación.
En el ejemplo de arriba, pais y anio son las columnas identificadoras y casos es la columna que contiene el valor de las observaciones.
En una tabla ancha, cada observación única se identifica a partir de la intersección de filas y columnas. En el ejemplo, los países están en las filas y los años en las columnas.
En general, el formato ancho es más compacto y legible por humanos mientras que el largo es más fácil de manejar con la computadora. Si revisás las tablas de arriba, es más fácil comparar los valores entre países y entre años en la tabla ancha. Pero el nombre de las columnas (“1999”, “2000”) en realidad ¡son datos! Además este formato se empieza a complicar en cuanto hay más de dos identificadores, como veremos más adelante.
Un mismo conjunto de datos puede ser representado de forma completamente “larga”, completamente “ancha” o –lo que es más común– en un formato intermedio. No existe una forma “correcta” de organizar los datos; cada una tiene sus ventajas y desventajas. Por esto es que es muy normal que durante un análisis los datos vayan y vuelvan entre distintos formatos dependiendo de los métodos estadísticos que se le aplican. Entonces, aprender a transformar datos anchos en largos y viceversa es un habilidad muy útil.
Desafío
En las tablas de ejemplo cada país tiene el un valor observado de “casos” para cada año. ¿Cómo agregarías una nueva variable con información sobre “precios”? Dibujá un esquema en papel y lápiz en formato ancho y uno en formato largo. ¿En qué formato es más “natural” esa extensión?
En esta sección vas a usar el paquete tidyr para manipular datos. Si no lo tenés instalado, podés hacerlo con el comando:
Cómo siempre, recordá que esto sólo se hace una vez y es recomendable hacerlo desde la consola para que no quede en un bloque de código por accidente.
Y luego cargá tidyr y dplyr (que usaste en una sección anterior) con:
7.1 De ancho a largo con pivot_longer()
En secciones anteriores usaste una versión de los datos asociados a parques nacionales. Ahora vas a leer los datos en su formato original:
parques_ancho <- readr::read_csv("http://datos.yvera.gob.ar/dataset/458bcbe1-855c-4bc3-a1c9-cd4e84fedbbc/resource/78aea6ed-761c-4659-bdf2-7fcb0f616fad/download/serie-tiempo-parques-nacionales-mensual.csv")
parques_ancho¿Notaste que en el código anterior no usaste library(readr) para cargar el paquete y luego leer?
Con la notación paquete::funcion() podés acceder a las funciones de un paquete sin tener que cargarlo.
Es una buena forma de no tener que cargar un montón de paquetes innecesarios si vas a correr una única función de un paquete pocas veces.
Esta tabla es bastante ancha y puede ser difícil de manejar. Por ejemplo, es complicado hacer series de tiempo de los visitantes de una región porque la información está distribuida entre varias columnas. Tampoco sería simple hacer cuentas por regiones o por tipo de residente con este formato de tabla.
Para convertirlo en una tabla más larga, se usa pivot_longer() (“longer” es “más largo” en inglés):
parques_largo <- parques_ancho %>%
pivot_longer(cols = -c("indice_tiempo", "residentes", "no_residentes", "total"),
names_to = "region_visitante",
values_to = "valor")
parques_largo## # A tibble: 2,952 × 6
## indice_tiempo residentes no_residentes total region_visitante valor
## <chr> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 2008-01 352303 198407 550710 buenos_aires_residentes 885
## 2 2008-01 352303 198407 550710 buenos_aires_no_residen… 0
## 3 2008-01 352303 198407 550710 buenos_aires_total 885
## 4 2008-01 352303 198407 550710 cordoba_residentes 717
## 5 2008-01 352303 198407 550710 cordoba_no_residentes 145
## 6 2008-01 352303 198407 550710 cordoba_total 862
## 7 2008-01 352303 198407 550710 cuyo_residentes 4965
## 8 2008-01 352303 198407 550710 cuyo_no_residentes 179
## 9 2008-01 352303 198407 550710 cuyo_total 5144
## 10 2008-01 352303 198407 550710 litoral_residentes 111408
## # … with 2,942 more rowsEl primer argumento depivot_longer() es la tabla que va a modificar: parques_ancho.
El segundo argumento se llama cols y es un vector con las columnas que tienen los valores a “alargar”.
Podría ser un vector escrito a mano (algo como c("buenos_aires_residentes", "buenos_aires_no_residentes"...)) pero con más de 20 columnas, escribir todo eso sería tedioso y probablemente estaría lleno de errores.
Por eso tidyr provee funciones de ayuda para seleccionar columnas en base a patrones.
Por ejemplo starts_with() que, como su nombre en inglés lo indica, selecciona las columnas que empiezan con una determinada cadena de caracteres.
en este caso le decimos que queremos todas las columnas menos las que están mencionadas. Por eso hay un “-” antes del vector de columnas.
Entonces, el vector -c("indice_tiempo", "residentes", "no_residentes", "total") le dice a pivot_longer() que seleccione todas las columnas excepto indice_tiempo, residentes, no_residentes y total.
Estas funciones accesorias para seleccionar muchas funciones se llaman “tidyselect”.
Si querés leer más detalles de las distintas formas que podés seleccionar variables leé la documentación usando ?tidyselect::language.
El tercer y cuarto argumento son los nombres de las columnas de “nombre” y de “valor” que va a tener la nueva tabla.
Como la nueva columna de identificación tiene los datos de la región y el tipo de visitante, region_visitante es un buen nombre.
Y la columna de valor va a tener… bueno, el valor.
Tomate un momento para visualizar lo que acaba de pasar. La tabla ancha tenía un montón de columnas con distintos datos. Ahora estos datos están uno arriba de otro en la columna “valor”, pero para identificar el nombre de la columna de la cual vinieron, se agrega la columna “region_visitante”.
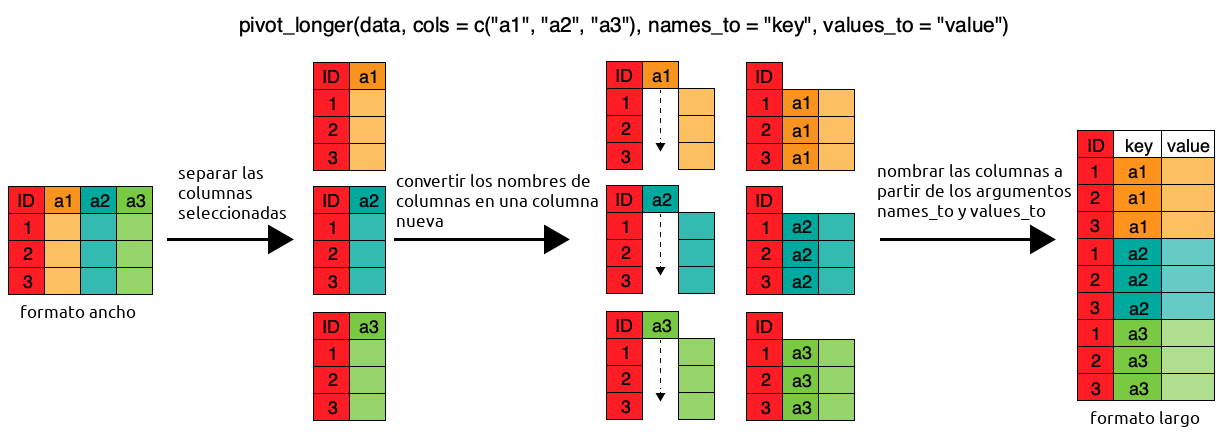
Proceso de ancho a largo
La columna region_visitante todavía no es muy útil porque contiene 2 datos, la región (Buenos Aires, Cuyo, Litoral, etc.) y el tipo de visitante (Residente, No Residente o la suma de ambos).
Sería mejor separar esta información en dos columnas llamadas “region” y “residente”.
Para eso está la función separate().
parques_largo <- parques_largo %>%
mutate(region_visitante = stringr::str_replace(region_visitante, "buenos_aires*", "buenos-aires"),
region_visitante = stringr::str_replace(region_visitante, "no_residentes", "noresidentes"))
separate(parques_largo,
region_visitante,
into = c("region", "tipo_visitante"),
sep = "_")## # A tibble: 2,952 × 7
## indice_tiempo residentes no_residentes total region tipo_visitante valor
## <chr> <dbl> <dbl> <dbl> <chr> <chr> <dbl>
## 1 2008-01 352303 198407 550710 buenos-a… residentes 885
## 2 2008-01 352303 198407 550710 buenos-a… noresidentes 0
## 3 2008-01 352303 198407 550710 buenos-a… total 885
## 4 2008-01 352303 198407 550710 cordoba residentes 717
## 5 2008-01 352303 198407 550710 cordoba noresidentes 145
## 6 2008-01 352303 198407 550710 cordoba total 862
## 7 2008-01 352303 198407 550710 cuyo residentes 4965
## 8 2008-01 352303 198407 550710 cuyo noresidentes 179
## 9 2008-01 352303 198407 550710 cuyo total 5144
## 10 2008-01 352303 198407 550710 litoral residentes 111408
## # … with 2,942 more rowsEl primer argumento, como siempre, es la tabla a procesar.
El segundo, col, es la columna a separar en dos (o más) columnas nuevas.
El tercero, into es el nombre de las nuevas columnas que separate() va a crear.
El último argumento es sep que define cómo realizar la separación.
Por defecto, sep es una expresión regular que captura cualquier caracter no alfanumérico.
En el caso de region_visitante no sirve, porque todos los valores que contienen, por ejemplo “buenos_aires_residentes” usan el “_” para separar palabras.
Si hubieamos usado la función separate() directamente hubieramos terminado con 3 columnas una con el datos “buenos”, otra con el datos “aires” y la última como el dato “residentes”.
Ni hablar si pensamos en “norte_residentes”, nos quedaríamos con 2 columnas.
Por esa razón primero tuvimos que manipular las columnas con mutate() para reemplazar “buenos_aires” por “buenos-aires” y “no_residentes” por “noresidentes” de manera de poder usar el “_” como separador entre la región y el tipo de visitante únicamente.
Así quedó la columna region_visitante antes de aplicar la separación.
parques_largo %>%
mutate(region_visitante = stringr::str_replace(region_visitante, "buenos_aires*", "buenos-aires"),
region_visitante = stringr::str_replace(region_visitante, "no_residentes", "noresidentes"))## # A tibble: 2,952 × 6
## indice_tiempo residentes no_residentes total region_visitante valor
## <chr> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 2008-01 352303 198407 550710 buenos-aires_residentes 885
## 2 2008-01 352303 198407 550710 buenos-aires_noresident… 0
## 3 2008-01 352303 198407 550710 buenos-aires_total 885
## 4 2008-01 352303 198407 550710 cordoba_residentes 717
## 5 2008-01 352303 198407 550710 cordoba_noresidentes 145
## 6 2008-01 352303 198407 550710 cordoba_total 862
## 7 2008-01 352303 198407 550710 cuyo_residentes 4965
## 8 2008-01 352303 198407 550710 cuyo_noresidentes 179
## 9 2008-01 352303 198407 550710 cuyo_total 5144
## 10 2008-01 352303 198407 550710 litoral_residentes 111408
## # … with 2,942 more rowsLa función str_replace() del paquete stringr busca un patrón en un texto, por ejemplo “no_residentes” y lo reemplaza por otra cadena de texto que indiquemos, por ejemplo “noresidentes”.
Este paquete es muy muy útil para manipular texto.
Desafío
Juntá todos los pasos anteriores en una sola cadena de operaciones usando %>%.
Nos falta eliminar esas columnas que ya no tienen sentido: residentes, no_resindetes y total. La información de esas columnas están asociadas al formato ancho y podemos volver a generarla si fuera necesario.
Guardemos el resultado de todos los pasos anteriores en parques_largo.
parques_largo <- parques_ancho %>%
pivot_longer(cols = -c("indice_tiempo", "residentes", "no_residentes", "total"),
names_to = "region_visitante",
values_to = "valor") %>%
mutate(region_visitante = stringr::str_replace(region_visitante, "buenos_aires*", "buenos-aires"),
region_visitante = stringr::str_replace(region_visitante, "no_residentes", "noresidentes")) %>%
separate(region_visitante, into = c("region", "tipo_visitante"), sep = "_") %>%
select(-c("residentes", "no_residentes", "total"))
parques_largo## # A tibble: 2,952 × 4
## indice_tiempo region tipo_visitante valor
## <chr> <chr> <chr> <dbl>
## 1 2008-01 buenos-aires residentes 885
## 2 2008-01 buenos-aires noresidentes 0
## 3 2008-01 buenos-aires total 885
## 4 2008-01 cordoba residentes 717
## 5 2008-01 cordoba noresidentes 145
## 6 2008-01 cordoba total 862
## 7 2008-01 cuyo residentes 4965
## 8 2008-01 cuyo noresidentes 179
## 9 2008-01 cuyo total 5144
## 10 2008-01 litoral residentes 111408
## # … with 2,942 more rowsSi decidimos que este es el formato ideal podríamos guardar los datos en un nuevo archivo csv y luego trabajar directamente con esa versión. Por supuesto, es importante guardar el código que lo genera pero eso puede ir en un archivo separado y se corre una única vez.
7.2 De largo a ancho con pivot_wider()
Ahora la variable parques_largo está en el formato más largo posible.
Tiene 4 columnas, de las cuales sólo una es la columnas con valores.
Pero con los datos así no podrías hacer un gráfico de puntos que muestre la relación entre cantidad de visitantes residentes y no residentes en cada mes como en la sección de gráficos.
Muchas veces es conveniente y natural tener los datos en un formato intermedio en donde hay múltiples columnas con los valores de distintas variables observadas.
Pasa “ensanchar” una tabla está la función pivot_wider() (“wider” es “más ancha” en inglés) y el código para conseguir este formato intermedio es:
parques_medio <- pivot_wider(parques_largo,
names_from = tipo_visitante,
values_from = valor)
parques_medio## # A tibble: 984 × 5
## indice_tiempo region residentes noresidentes total
## <chr> <chr> <dbl> <dbl> <dbl>
## 1 2008-01 buenos-aires 885 0 885
## 2 2008-01 cordoba 717 145 862
## 3 2008-01 cuyo 4965 179 5144
## 4 2008-01 litoral 111408 55335 166743
## 5 2008-01 norte 4241 774 5016
## 6 2008-01 patagonia 230087 141973 372060
## 7 2008-02 buenos-aires 624 0 624
## 8 2008-02 cordoba 475 148 623
## 9 2008-02 cuyo 4803 139 4942
## 10 2008-02 litoral 85853 53596 139449
## # … with 974 more rowsNuevamente el primer argumento es la tabla original.
El segundo, names_from es la columna cuyos valores únicos van a convertirse en nuevas columnas.
La columna tipo_visitante tiene los valores "residentes", "noresidentes" y "total" y entonces la tabla nueva tendrá tres columnas con esos nombres.
El tercer argumento, values_from, es la columna de la cual sacar los valores.
Para volver al formato más ancho, basta con agregar más columnas en el argumento names_from:
pivot_wider(parques_largo,
names_from = c(region, tipo_visitante),
names_sep = "_",
values_from = valor)## # A tibble: 164 × 19
## indice_tiempo `buenos-aires_residentes` `buenos-aires_nore… `buenos-aires_to…
## <chr> <dbl> <dbl> <dbl>
## 1 2008-01 885 0 885
## 2 2008-02 624 0 624
## 3 2008-03 0 0 0
## 4 2008-04 462 0 462
## 5 2008-05 1091 0 1091
## 6 2008-06 1126 0 1126
## 7 2008-07 1304 0 1304
## 8 2008-08 2912 0 2912
## 9 2008-09 1394 0 1394
## 10 2008-10 2004 0 2004
## # … with 154 more rows, and 15 more variables: cordoba_residentes <dbl>,
## # cordoba_noresidentes <dbl>, cordoba_total <dbl>, cuyo_residentes <dbl>,
## # cuyo_noresidentes <dbl>, cuyo_total <dbl>, litoral_residentes <dbl>,
## # litoral_noresidentes <dbl>, litoral_total <dbl>, norte_residentes <dbl>,
## # norte_noresidentes <dbl>, norte_total <dbl>, patagonia_residentes <dbl>,
## # patagonia_noresidentes <dbl>, patagonia_total <dbl>En esta llamada también está el argumento names_sep, que determina el caracter que se usa para crear el nombre de las nuevas columnas, usamos "_" para que quede igual al original.
Desafío
- ¿Cómo es la tabla más ancha posible que podés generar con estos datos? ¿Cuántas filas y columnas tiene? No es necesario que lo intentes hacer ahora pero siempre sirve hacer un diagrama para organizar las ideas.
7.3 Uniendo tablas
Hasta ahora todo lo que usaste de dplyr involucra trabajar y modificar con una sola tabla a la vez, pero es muy común tener dos o más tablas con datos relacionados.
En ese caso, tenemos que unir estas tablas a partir de una o más variables en común o keys.
En Excel u otro programa de hojas de cálculo, esto se resuelve con la función “VLOOKUP” o “BUSCARV”, en R y en particular dentro del mundo de dplyr hay que usar la familia de funciones *_join().
Hay una función para cada tipo de unión que queramos hacer.
Asumiendo que querés unir dos data.frames o tablas x e y que tienen en común una variable A:
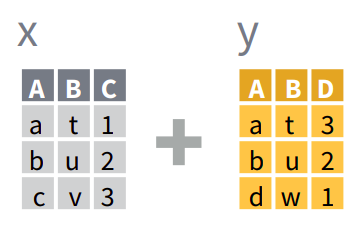
full_join(): devuelve todas las filas y todas las columnas de ambas tablasxey. Cuando no coinciden los elementos eny, devuelveNA(dato faltante). Esto significa que no se pierden filas de ninguna de las dos tablas aún cuando no hay coincidencia. Está es la manera más segura de unir tablas.left_join(): devuelve todas las filas dexy todas las columnas dexey. Las filas enxque no tengan coincidencia conytendránNAen las nuevas columnas. Si hay múltiples coincidencias entrexey, devuelve todas las coincidencias posibles.right_join(): es igual queleft_join()pero intercambiando el orden dexey. En otras palabras,right_join(x, y)es idéntico aleft_join(y, x).inner_join(): devuelve todas las filas dexdonde hay coincidencias conyy todas las columnas dexey. Si hay múltiples coincidencias entrexey, entonces devuelve todas las coincidencias. Esto significa que eliminará las filas (observaciones) que no coincidan en ambas tablas, lo que puede ser peligroso.

Familia de uniones de dplyr
Ahora vamos a seguir trabajando con las base de datos de parques_largo y de paso unirlo a una nueva base de datos hoteles que contiene información de viajeros por región de destino y origen a lo largo del tiempo.
Para que los datos sean más manejables y poder ver que es lo que sucede vamos a quedarnos sólo con la información de 2018.
library(lubridate)
library(stringr)
parques_2018 <- parques_largo %>%
mutate(indice_tiempo = ym(indice_tiempo)) %>%
filter(year(indice_tiempo) == 2018)
hoteles <- readr::read_csv("http://datos.yvera.gob.ar/dataset/93db331e-6970-4d74-8589-c1223ac9c282/resource/1f6b78aa-d3b4-440a-bd7d-30d76e1728aa/download/viajeros-hospedados-residentes-y-no-residentes-por-destino.csv")
hoteles_2018 <- hoteles %>%
mutate(region_de_destino = tolower(region_de_destino) %>%
str_replace(" ", "-") %>%
str_replace("ó", "o")) %>%
pivot_wider(names_from = origen_viajeros, values_from = viajeros) %>%
rename(hoteles_noresidentes = `No residentes`,
hoteles_residentes = Residentes,
region = region_de_destino) %>%
mutate(indice_tiempo = lubridate::ym(indice_tiempo)) %>%
filter(lubridate::year(indice_tiempo) == 2018)En el código de más arriba volvimos a usar la librería lubridate.
En este caso la función year() permite extraer el parte del año de la fecha que está guardada en la columna indice_tiempo.
Esto luego nos pemite filtrar los datos por años.
Esta nueva tabla tiene 5 columnas: indice_tiempo con la fecha (año y mes), region con la regiones de destino de los visitantes, observaciones con muchos NA, hoteles_residentes y hoteles_noresidentes que indica la cantidad de personas resindetes y no residentes que se alojaron en hoteles.
Las columnas region e indice_tiempo también están presente en la tabla parques_2018 y son las que van a servir como variables llave para unir las dos tablas.
Para unir las dos tablas, cualquier función join requiere cierta información:
- las tablas a unir: son los dos primeros argumentos.
- qué variable o variables (se puede usar más de una!) usar para identificar coincidencias: el argumento
by.
Unamos parques_2018 y hoteles_2018 primero con full_join():
parques_hoteles_2018 <- full_join(parques_2018, hoteles_2018,
by = c("region", "indice_tiempo"))
parques_hoteles_2018## # A tibble: 228 × 7
## indice_tiempo region tipo_visitante valor observaciones hoteles_residen…
## <date> <chr> <chr> <dbl> <chr> <dbl>
## 1 2018-01-01 buenos-aires residentes 239 <NA> 385396
## 2 2018-01-01 buenos-aires noresidentes 4 <NA> 385396
## 3 2018-01-01 buenos-aires total 243 <NA> 385396
## 4 2018-01-01 cordoba residentes 2663 <NA> 299440
## 5 2018-01-01 cordoba noresidentes 239 <NA> 299440
## 6 2018-01-01 cordoba total 2902 <NA> 299440
## 7 2018-01-01 cuyo residentes 4959 <NA> 141927
## 8 2018-01-01 cuyo noresidentes 259 <NA> 141927
## 9 2018-01-01 cuyo total 5218 <NA> 141927
## 10 2018-01-01 litoral residentes 157509 <NA> 261555
## # … with 218 more rows, and 1 more variable: hoteles_noresidentes <dbl>Si miramos de cerca la tabla unida veremos un par de cosas:
- Todas las columnas de
parques_2018y dehoteles2018están presentes. - Todas las observaciones están presentes, aún las regiones que están presentes en
hoteles_2018pero no enparques_2018(no hay parques nacionales en CABA). En esos casos ahora tenemosNAen las columnas que vienen del data.frame de parques. Esto genera una tabla con 228 filas.
Esta es la opción más segura si no sabemos si todas las observaciones de una tabla están presente en a otra.
Si solo nos interesa conservar las filas de la tabla de la izquierda, en este caso parques_2018 entonces:
parques_hoteles_2018 <- left_join(parques_2018, hoteles_2018,
by = c("region", "indice_tiempo"))
parques_hoteles_2018## # A tibble: 216 × 7
## indice_tiempo region tipo_visitante valor observaciones hoteles_residen…
## <date> <chr> <chr> <dbl> <chr> <dbl>
## 1 2018-01-01 buenos-aires residentes 239 <NA> 385396
## 2 2018-01-01 buenos-aires noresidentes 4 <NA> 385396
## 3 2018-01-01 buenos-aires total 243 <NA> 385396
## 4 2018-01-01 cordoba residentes 2663 <NA> 299440
## 5 2018-01-01 cordoba noresidentes 239 <NA> 299440
## 6 2018-01-01 cordoba total 2902 <NA> 299440
## 7 2018-01-01 cuyo residentes 4959 <NA> 141927
## 8 2018-01-01 cuyo noresidentes 259 <NA> 141927
## 9 2018-01-01 cuyo total 5218 <NA> 141927
## 10 2018-01-01 litoral residentes 157509 <NA> 261555
## # … with 206 more rows, and 1 more variable: hoteles_noresidentes <dbl>Ahora esperamos que la tabla resultante tenga la misma cantidad de filas que parques_2018 y efectivamente eso ocurre.
Todas las regiones en parques_2018 tienen coincidencia con hoteles_2018 y por eso no hay NAs en las columnas que vienen de ese último data.frame.
Finalmente, si quisiéramos quedarnos solo con las observaciones que están presentes en ambas tablas usamos inner_join().
parques_hoteles_2018 <- inner_join(parques_2018, hoteles_2018,
by = c("region", "indice_tiempo"))
parques_hoteles_2018## # A tibble: 216 × 7
## indice_tiempo region tipo_visitante valor observaciones hoteles_residen…
## <date> <chr> <chr> <dbl> <chr> <dbl>
## 1 2018-01-01 buenos-aires residentes 239 <NA> 385396
## 2 2018-01-01 buenos-aires noresidentes 4 <NA> 385396
## 3 2018-01-01 buenos-aires total 243 <NA> 385396
## 4 2018-01-01 cordoba residentes 2663 <NA> 299440
## 5 2018-01-01 cordoba noresidentes 239 <NA> 299440
## 6 2018-01-01 cordoba total 2902 <NA> 299440
## 7 2018-01-01 cuyo residentes 4959 <NA> 141927
## 8 2018-01-01 cuyo noresidentes 259 <NA> 141927
## 9 2018-01-01 cuyo total 5218 <NA> 141927
## 10 2018-01-01 litoral residentes 157509 <NA> 261555
## # … with 206 more rows, and 1 more variable: hoteles_noresidentes <dbl>En este caso, perdemos las filas de hoteles_2018 que no encontraron coincidencia en parques_2018, es decir la región de CABA.
La tabla resultante es igual a la tabla que generamos en el ejemplo anterior con 216 filas.
Desafío
Ahora es tu turno.
Nos faltó revisar la función right_join().
Intentá unir las dos tablas con las que estuvimos trabajando cambiando el orden en el que las llamas en la función.
- ¿Ves alguna diferencia en los resultados?
- Si encontras alguna diferencia, intentá explicar a que se debe. Si no hay diferencias, pensá por qué.
Hasta ahora en las uniones usamos columnas que tenian el mismo nombre en ambas tablas. ¿Qué ocurre cuando las columnas no se llaman igual?. Una solución es renombrar las columnas en alguna de la tablas (como en los ejemplos). La otra opción es indicar como se llaman las columnas que tenemos que unir en el parámetro by de la función join. Por ejemplo, si en la tabla parques region se llama region y en la tabla hoteles se llama regiones, el parámetro by debe ser especificado como: by = c("region" = "regiones")